Machado, C. R. D., M. Azambuja, C. Domit, G. Fraga da Fonseca, L. Glugoski, C. Borges Gazolla, R. Bonfim de Almeida, M. B. Pucci, T. Torres Pires, V. Nogaroto & M. R. Vicari (2023): Integrating morphological, molecular and cytogenetic data for F2 sea turtle hybrids diagnosis revealed balanced chromosomal sets. – Journal of Evolutionary Biology 36(11): 1595–1608.
Integrierte morphologische, molekulare und zytogenetische Daten für die F2 Meeresschildkröten-Hybriddiagnosen zeigen ausbalancierte chromosomale Anlagen.
DOI: 10.1111/jeb.14236 ➚

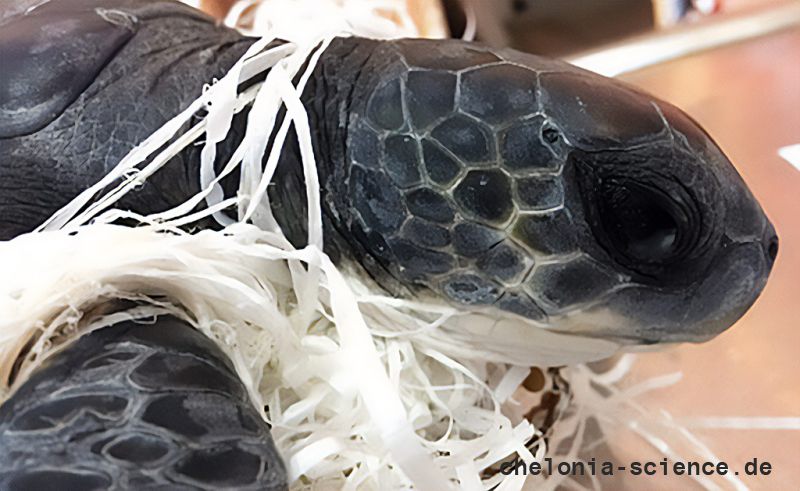
aus Caretta caretta × Eretmochelys
imbricata × Chelonia mydas
© Caroline Regina Dias Machado
Hybridisierung kann ein Teil der Evolutionsgeschichte bei vielen Spezies sein. Die Hybridisierung zwischen Meeresschildkrötenarten entlang der brasilianischen Küste erscheint atypisch und tritt dort auf, wo sich die Nistareale und die Reproduktionssaison überschneiden. Die integrierte Untersuchung bezogen auf die Morphologie und die Genetik sind immer noch selten und somit gibt es kaum Nachweise für die Verteilung der elterlichen Chromosomensätze für die Meeresschildkrötenhybriden. Bei dieser Studie kombinierten wir Chromosomenmarker die schon früher für artreine Meeresschildkrötenspezies etabliert worden waren mit morphologischen und molekularen Analysen mit dem Ziel damit die genetische Zusammensetzung des Chromosomensatzes bei den möglichen Interspezieshybriden, die aufgrund ihrer gemischten Morphologie identifiziert worden waren, zu analysieren. Die Ergebnisse zeigten, dass eines der Hybridindividuen ein F2-Hybridindividuum hervorgegangen aus Caretta caretta × Eretmochelys imbricata × Chelonia mydas sein könnte und ein weiteres könnte aus einer Rückkreuzung zwischen C. caretta × Lepidochelys olivacea abstammen. Die natürlichen Allele mit verschiedener Herkunft aus den elterlichen Linien konnten in den Hybriden gezeigt werden und es konnte bewiesen werden, dass diese Hybrid-Chromosomensätze trotzdem in ausbalancierter Form vorlagen. Wie diese Meeresschildkrötenhybridisierung sich langfristig auf die genetischen Parameter auswirken wird bleibt bedenklich, da die Auswirkungen dieser Meeresschildkrötenhybridisierungen auf das Crossing-over bei den Hybridchromosomensätzen und deren genetisch-funktionelle Konsequenzen bislang unvorhersagbar sind.
Kommentar von H.-J. Bidmon
Der letzte Satz dieses Abstracts ist zwar richtig, aber ich denke, wenn wir uns einmal die verschiedensten Evolutionslinienentwicklungen bei den Schildkröten und auch bei anderen Spezies anschauen, dann halte ich diese Bedenken für eines der geringsten Probleme mit denen die Aufrechterhaltung von Schildkrötenevolutionslinien zu kämpfen hat, denn wir sehen wie bei unserer eigenen menschlichen Evolutionsline, dass sie viele solcher Ereignisse mehr oder weniger problemlos überdauert haben. Letztendlich könnte man fast davon ausgehen, dass wie bei der Biodiversitätsevaluation höhere Diversitäten eine bessere Ökosystemstabilität indizieren könnte, so könnte auch eine Erhöhung der genetischen Diversität durch Hybridisierung zur Bestandserhaltung langfristig beitragen. Sicher werden jetzt viele die Sachlage so interpretieren, dass bei der Hybridisierung aus zwei Arten eine hervorgeht und dadurch Diversität verloren geht, aber ich glaube, wenn wir in der biologischen Evolution einmal zurückblicken verwechseln wir da etwas Gravierendes was eben auch zum Evolutionslinienerhalt und später dann auch zur Wiederaufspaltung in neue Linien einen wesentlichen Beitrag geleistet hat. Damit schließe ich gar nicht aus, dass Hybridisierung auch tödliche Nachteile in dem einen oder anderen Fall nach sich ziehen kann, aber solche, wie uns die Biologie nur allzu oft gezeigt hat, sind solche Evolutionsverläufe kontextabhängig und die Umweltbedingungen und Umweltveränderungen liefern nun mal vielfältige Kontexte oder Szenarien und deshalb sollten wir nicht vergessen, dass selbst wir ein Produkt solch einer retikulierten Evolution sind, da auch wir Neandertaler- und/oder Denisovagenomanteile aufweisen (Harris et al., 2023; Velazquez-Arcelay et al., 2023; siehe dazu auch Renner, 2016, Bidmon, 2017; Hennessy, 2020; Arantes et al., 2020; Vilaça et al., 2022) und die dortigen Kommentare). Für all diejenigen die sich weitergehend mit dieser Thematik beschäftigen möchten, empfehle ich die an einem Modellorganismus, dem Schwerträgern aus der Gattung Xiphophorus, erst jüngst detaillierter erarbeiteten Daten (Du et al., 2024; Moran et al., 2024; Langdon et al., 2023). Gerade bei der Betrachtung dieser Arbeiten wird auch deutlich, wie oft außerhalb der Wissenschaft häufig nur selektiv diskutiert wird. Denn, wenn man sich nur auf die Arbeit mit dem Titel: „A lethal mitonuclear incompatibility in complex I of natural hybrids“ fokussiert ist Hybridisierung sicherlich ein Todesurteil, aber wie die beiden anderen Arbeiten (Du et al., 2024; Langdon et al., 2023) zeigen hält die „Belebte Materie“ auch Auswege offen und letztere scheinen bei langfristiger Betrachtung die bedeutendere Rolle zu spielen. Ob uns diese Erkenntnisse auch dazu veranlassen, sollten wir den Begriff Art bzw. Spezies neu zu definieren, bleibt offen. Wir sollten aber immer im Blick behalten, dass menschengemachte, abstrakte Definitionen immer nur das Wissen für die Entscheidungsfindung und Begründung nutzen können welches uns derzeit bekannt ist - dieses Wissen muss aber nicht der Realität entsprechen, denn wie schon öfters erwähnt Realität beruht auch auf dem was wir noch nicht Wissen und somit auch noch nicht mit den besten Computerprogrammen voraussagen oder gar langfristig realitätsnah modulieren könnten.
Literatur
Arantes, L. S., L. C. L. Ferreira, M. Driller, F. P. M. Repinaldo Filho, C. J. Mazzoni & F. R. Santos (2020): Genomic evidence of recent hybridization between sea turtles at Abrolhos Archipelago and its association to low reproductive output. – Scientific Reports 10(1): 12847 oder Abstract-Archiv.
Bidmon, H.-J. (2017): Sind phylogenetische Stammbäume nur ein Traum? – Schildkröten im Fokus 14(1): 14-27 ➚.
Du, K., Y. Lu, M. Garcia-Olazabal, R. B. Walter, W. C. Warren, T. Dodge, M. Schumer, H. Park, A. Meyer & M. Schartl (2024): Phylogenomics analyses of all species of Swordtails (Genus Xiphophorus) highlights hybridization precedes speciation. – bioRxiv 2023.12.30.573732; DOI: 10.1101/2023.12.30.573732 ➚.
Harris, D. N., A. Platt, M. E. B. Hansen, S. Fan, M. A. McQuillan, T. Nyambo, S. W. Mpoloka, G. G. Mokone, G. Belay, C. Fokunang, A. K. Njamnshi & S. A. Tishkoff (2023): Diverse African genomes reveal selection on ancient modern human introgressions in Neanderthals – Current biology 33(22): 4905-4916.e5; DOI: 10.1016/j.cub.2023.09.066 ➚.
Hennessy, E. (2020): Saving Species: The Co-Evolution of Tortoise Taxonomy and Conservation in the Galápagos Islands. – Environmental History 25(2): 263-286 oder Abstract-Archiv.
Langdon QK, Groh JS, Aguillon SM, Powell DL, Gunn T, Payne C, Baczenas JJ, Donny A, Dodge TO, Du K, Schartl M, Ríos-Cárdenas O, Gutierrez-Rodríguez C, Morris M, Schumer (2023): Genome evolution is surprisingly predictable after initial hybridization. – bioRxiv 2023.12.21.572897; DOI: 10.1101/2023.12.21.572897 ➚.
Moran, B. M., C. Y. Payne, D. L. Powell, E. N. K. Iverson, A. E. Donny, S. M. Banerjee, Q. K. Langdon, T. R. Gunn, R. A. Rodriguez-Soto, A. Madero, J. J. Baczenas, K. M. Kleczko, F. Liu, R. Matney, K. Singhal, R. D. Leib, O. Hernandez-Perez, R. Corbett-Detig, J. Frydman, C. Gifford, M. Schartl, J. C. Havird & M. Schumer (2024): A lethal mitonuclear incompatibility in complex I of natural hybrids. – Nature 626(7997): 119-127; DOI: 10.1038/s41586-023-06895-8 ➚.
Renner, S. S. (2016): A Return to Linnaeus's Focus on Diagnosis, Not Description: The Use of DNA Characters in the Formal Naming of Species. – Systematic Biology 65(6): 1085-1095 oder Abstract-Archiv.
Velazquez-Arcelay, K., L. L. Colbran, E. McArthur, C. M. Brand, D. C. Rinker, J. K. Siemann, D. G. McMahon & J. A. Capra (2023): Archaic Introgression Shaped Human Circadian Traits. – Genome Biology and Evolution 15(12): evad203; DOI: 10.1093/gbe/evad203 ➚.
Vilaça, S. T., F. Maroso, P. Lara, B. de Thoisy, D. Chevallier, L. Souza Arantes, F. R. Santos, G. Bertorelle, C. J. Mazzoni (2022): Evidence of backcross inviability and mitochondrial DNA paternal leakage in sea turtle hybrids. – Molecular Ecology 32(3): 628-643 oder Abstract-Archiv.
Galerien








Caretta caretta – Unechte Karettschildkröte





Chelonia mydas – Grüne Meeresschildkröte

Eretmochelys imbricata – Echte Karettschildkröte




Hybrid Caretta caretta x Eretmochelys imbricata x Chelonia mydas



Lepidochelys olivacea – Oliv-Bastardschildkröte